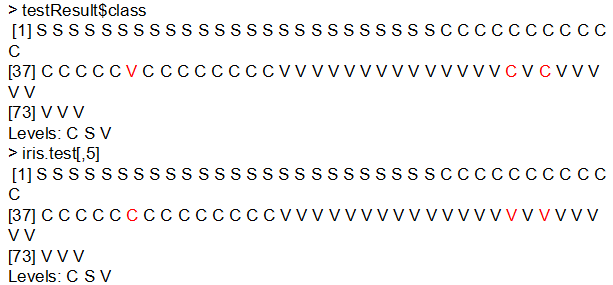【Rによるデータサイエンス】線形判別分析
◎本題に入る前に1
余談・・・。
最近、徐々に内容が難しくなってきていて、自分の予備知識の足りなさを痛感している。ただ、今回の「Rによるデータサイエンス」の初読の目的は「やった感+写経による学習+概要を把握すること」なので、わからないことがあっても基本的には飛ばすし、深入りもしない。
◎本題に入る前に2
本で述べられていることだけど、判別分析は最も古典的なパターン認識の手法の1つらしい。
で、パターン認識が何者なのかを私は知らないのだが、本には「識別・認識に関することを機械的に実現する研究分野」とある。
それでもよく分からないからググっていたら次の資料に遭遇。読み物として面白いので載せておく。
この資料には次のようにある。
- 認識とは、不要な情報を捨てること
- 特徴選択・特徴抽出が識別・認識の本質
これだけでだいぶ満足というか興奮してしまったw。もっとも最後まで読んだからこそ、そう思えるのかもしれないが。
また、全部読んで思ったが、この資料の作成者は、本当にパターン認識と機械学習を体得しているんだなぁと。
さて、前置きが長くなったが、いつもどおりの体裁でここから学習。
◎本題
【定義】
判別分析とは・・・。
判別分析とは、個体がどのグループに属するかが明確である学習データを用いて判別モデルを構築し、そのモデルを用いて所属不明の個体(テスト用のデータ)がどのグループに帰属するかを判別する方法。
ふーん。そうであれば、判別分析を実施するにあたり、次の2つが必要ということだろうか。
【必要なもの1】個体がどのグループに属するかが明確である学習データ
【必要なもの2】判別モデル
そういうことなら、「【必要なもの1】」として、Rにはirisデータが用意されている。
> iris[c(1:3,51:52),] Sepal.Length Sepal.Width Petal.Length Petal.Width Species 1 5.1 3.5 1.4 0.2 setosa 2 4.9 3.0 1.4 0.2 setosa 3 4.7 3.2 1.3 0.2 setosa 51 7.0 3.2 4.7 1.4 versicolor 52 6.4 3.2 4.5 1.5 versicolor
最後の列のSpecies列が各個体のグループ情報、つまり外的規準となる。
これを見て分かる通り、外的規準Speciesの値は「setosa」と「versicolor」という属の種類である。つまり、判別分析において、外的規準は定量的データではなく、定性的データである。
これで「【必要なもの1】個体がどのグループに属するかが明確である学習データ」は用意出来た。次は、「【必要なもの2】判別モデル」。
これには、「Fisher判別分析」というものがあるらしい。
これは、データが多変量正規分布に従い、グループの母分散が等しいという仮定のもとで、群間の分散と郡内の分散の比を最大化することで判別関数の係数を求める方法。
はい、さっぱりですw。
この資料に背景が書いている。
◎ケーススタディ
Rに標準で用意されているirisデータを使用して、3群判別分析を行う。
3群判別分析とは、所属不明の個体が3つのグループのいずれに属するかを判別することである。
なんで3つかは、次のように、今回は既知データで3種であることがわかっているからだろう。なので、じゃっかんインチキ?
> unique(iris[,5:5]) #irisのSpeciesの種類を抜き出す [1] setosa versicolor virginica Levels: setosa versicolor virginica
まず、データを準備する。
学習データiris.trainとテストデータiris.testに分けて作成する。
> iris.lab<-c(rep("S",50),rep("C",50),rep("V",50)) > iris1<-data.frame(iris[,1:4],Species=iris.lab) > even.n<-2*(1:75)-1 > iris.train<-iris1[even.n,] > iris.test<-iris1[-even.n,]
線形判別関数ldaを実行する。
lda(formula, data)
formulaには、「グループの識別変数~変数」のように記述する。
> library(MASS) > (Z.lda<-lda(Species~.,data=iris.train)) Call: lda(Species ~ ., data = iris.train) Prior probabilities of groups: C S V 0.3333333 0.3333333 0.3333333 Group means: Sepal.Length Sepal.Width Petal.Length Petal.Width C 5.992 2.776 4.308 1.352 S 5.024 3.480 1.456 0.228 V 6.504 2.936 5.564 2.076 Coefficients of linear discriminants LD1 LD2 Sepal.Length -0.5917846 -0.1971830 Sepal.Width -1.8415262 2.2903417 Petal.Length 1.6530521 -0.7406709 Petal.Width 3.5634683 2.6365924 Proportion of trace: LD1 LD2 0.9913 0.0087
返却された線形判別係数(coefficients of linear discriminants)を用いて判別関数を構築する。
判別関数は用いる変数と判別係数との線形結合である。
この結果を見ると2つの判別係数(LD1、LD2)が返却されている。
LD1を使用すると判別関数は次のようになる。
ここでCは定数項で、グループの平均と判別係数との線形結合の平均値である。
グループの平均は次のように計算できる。
> Z.lda$means Sepal.Length Sepal.Width Petal.Length Petal.Width C 5.992 2.776 4.308 1.352 S 5.024 3.480 1.456 0.228 V 6.504 2.936 5.564 2.076
判別係数は次のように計算できる。
> Z.lda$scaling LD1 LD2 Sepal.Length -0.5917846 -0.1971830 Sepal.Width -1.8415262 2.2903417 Petal.Length 1.6530521 -0.7406709 Petal.Width 3.5634683 2.6365924
これら2つから「グループの平均」と「判別係数」の線形結合は、コマンド%*%で行列の積演算を実施することで次のように求められる。
> Z.lda$means%*%Z.lda$scaling LD1 LD2 C 3.281108 5.550331 S -6.162322 6.502468 V 7.339654 6.794438
さらにapply関数を使用してLD1とLD2の列ごとに平均をとることで、「グループの平均と判別係数との線形結合の平均値」である定数項Cを求めると次のようになる*1。
> apply(Z.lda$means%*%Z.lda$scaling,2,mean) LD1 LD2 1.486146 6.282412
◎学習の結果と判別
判別結果を関数predictを用いて表そうと思ったが・・・。
> table(iris.train[,5],predict(Z.lda)$class) 以下にエラー UseMethod("predict") : 'predict' をクラス "lda" のオブジェクトに適用できるようなメソッドがありません
エラー!なんで?
何回やってもうまくいかないなぁ。おかしいなぁ。と思いながら、ldaを実行すると・・・。
> (test<-lda(Species~ .,data=train.data)) エラー: 関数 "lda" を見つけることができませんでした
いつの間にか、ワークスペース削除してた?ようだ・・・。眠かったからなぁ。。。
そういうわけで、library(MASS)の実行も含めて最初からやり直してldaを実行すると次のように成功。
> table(iris.train[,5],predict(Z.lda)$class) C S V C 24 0 1 S 0 25 0 V 1 0 24
この結果によると、Cグループのうち1つがVグループに、Vグループのうち1つがCグループに分類されている(誤判別)。
判別関数得点をグラフにしてみると。
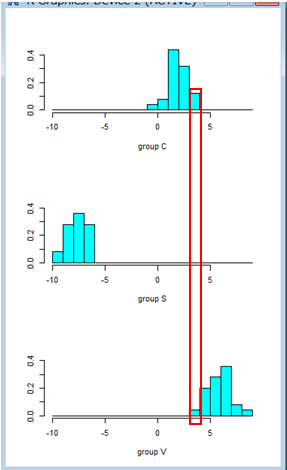
> plot(Z.lda,dimen=1)
group Cとgroup Vのグラフが重なっており、誤判別を確認することができる。
このグラフ、横軸が判別関数得点で、縦軸が個数だと思っていたが、なんで縦軸の値が小数なの???
ひとまず気にせず、次はdimen=2として判別関数得点の散布図を描いてみる。
> plot(Z.lda,dimen=2)
これで、横軸が第1判別関数の値、縦軸が第2判別関数の値である散布図が作成される。
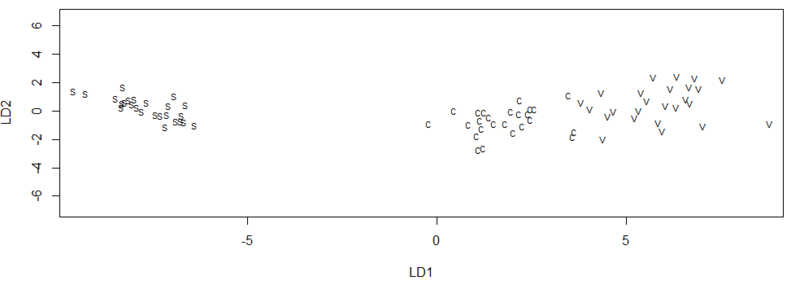
なお、第1判別関数の値というのは、第1判別関数の係数LD1を使用した場合に計算される判別関数式の得点(判別関数得点)。
関数lda実行結果のpredict$xで求められる。
> predict(Z.lda)$x LD1 LD2 1 -7.9226230 0.21852960 3 -7.2987565 -0.31563262 5 -8.0475972 0.46728206 7 -7.0862313 0.35174617 9 -6.4034581 -1.01764732 11 -8.3031584 0.54337595 13 -7.1806714 -1.13114559
◎テストデータの判別
ここまでは、学習用データiris.trainに対して線形判別ldaを実施し、その結果をZ.ldaに格納した。
> (Z.lda<-lda(Species~.,data=iris.train)) Call: lda(Species ~ ., data = iris.train) Prior probabilities of groups: C S V 0.3333333 0.3333333 0.3333333 Group means: Sepal.Length Sepal.Width Petal.Length Petal.Width C 5.992 2.776 4.308 1.352 S 5.024 3.480 1.456 0.228 V 6.504 2.936 5.564 2.076 Coefficients of linear discriminants: LD1 LD2 Sepal.Length -0.5917846 -0.1971830 Sepal.Width -1.8415262 2.2903417 Petal.Length 1.6530521 -0.7406709 Petal.Width 3.5634683 2.6365924 Proportion of trace: LD1 LD2 0.9913 0.0087
次は、求められた判別関数を使用してテストデータについて判別分析を行う。
分析の流れは次の通り。
関数predictを使用して、学習データから求めた判別関数を使用して、testデータの判別結果を求める。
> testResult<-predict(Z.lda,iris.test[,-5])
テストデータのグループラベルと、テストデータから求めた判別結果のグループラベルのクロス表を作成して、判別の精度を確認する。
> table(iris.test[,5],testResult$class) C S V C 24 0 1 S 0 25 0 V 2 0 23
この結果を見ると、Cグループの1つがVに、Vグループの2つがCに誤判別されている。
実際にデータを確認するとよく分かる。
> testResult$class [1] S S S S S S S S S S S S S S S S S S S S S S S S S C C C C C C C C C C C [37] C C C C C V C C C C C C C C V V V V V V V V V V V V V V C V C V V V V V [73] V V V Levels: C S V > iris.test[,5] [1] S S S S S S S S S S S S S S S S S S S S S S S S S C C C C C C C C C C C [37] C C C C C C C C C C C C C C V V V V V V V V V V V V V V V V V V V V V V [73] V V V Levels: C S V
可視化する。
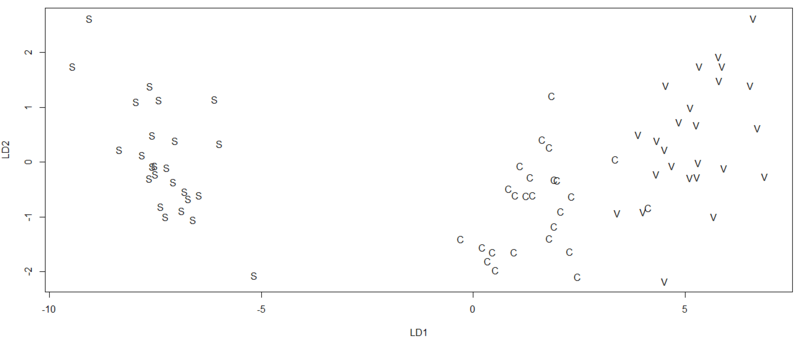
> plot(testResult$x,type="n") > text(testResult$x,labels=iris.test$Species)
*1:関数applyの第2引数に2を指定すると、第1引数の行列の列ベクトルごとに、第3引数の関数を適用(apply)することになる。